基本介绍
在上周的文章中,我们带大家走进了单细胞测序的世界,对单细胞测序有了初步了解。今天我们将帮大家进一步熟悉单细胞转录组测序(Single Cell RNA sequencing,scRNA-seq),回顾它的发展历史,技术进程及应用案例。
单细胞测序技术首次由Tang et al. 于2009年发表,用以比较单细胞测序和微芯片技术。2011年Islam et al.创建了第一个复用scRNA测序库,它的规模和通量的增加为3年后scRNA-seq的广泛普及奠定了基础。新平台只需两步就可以使单细胞分离和文库生成,它的出现改变了单细胞测序成本高、人力需求高且protocols有限的现状。drop-seq技术也紧接着在2015年问世,它允许将一个细胞和一个功能珠压缩到油乳剂中的一个液滴中,使得细胞裂解、条形码和反转录可以在单个液滴中完成。第一个便携式单细胞文库生成平台,称为seq-well的单细胞库制备程序也在2017年由Gierahn et al.开发。近几年,scRNA-seq出现了爆发式的发展和普及,技术不断优化和创新,可以检测到更多的细胞,更高的分辨率,更全面的信息。
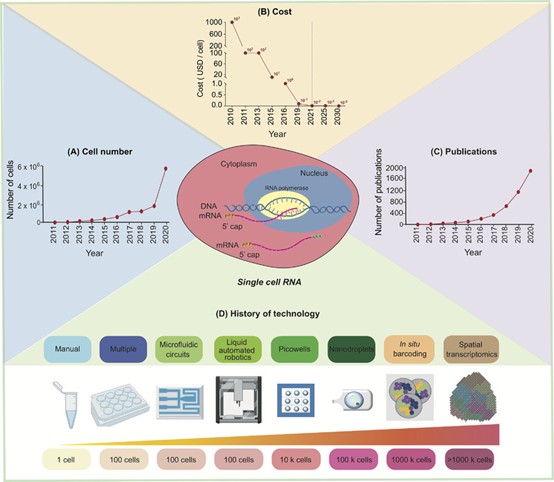
图1. 近十年单细胞测序爆发式的发展
技术原理
scRNA-seq的主要流程可以分为以下几个步骤:
制备单细胞悬液:从样本组织中提取并且制备单细胞悬液。
分离、捕获单细胞:常见的单细胞分离捕获技术包括荧光激活细胞分选 (FACS)、磁激活细胞分选、微流体系统和激光显微切割。
提取RNA:使用特定的试剂盒或技术将单个细胞中的RNA提取出来,进行质量检测和定量分析。
逆转录和扩增:使用逆转录酶将RNA转化为cDNA,通常通过PCR或者体外扩增(IVT)进行扩增,以增加信号强度和覆盖度。这个步骤中利用了UMI技术,将每个mRNA条形码化,以辨识细胞中的每个单独的mRNA。
构建文库和测序:使用特定的方法或平台将cDNA构建成文库,并进行高通量测序,以获得每个细胞的基因表达数据。
数据分析:使用专门的软件或算法对测序数据进行质控、标准化、降维、聚类、差异分析等,以揭示单细胞水平的基因表达模式和功能。
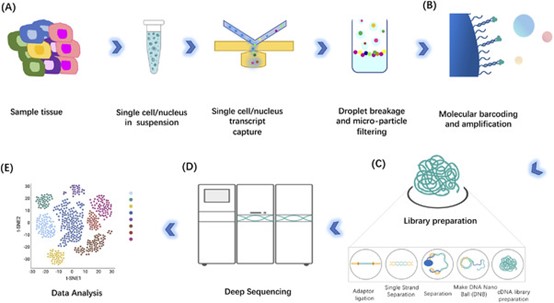
图2. 单细胞测序的步骤
在单细胞测序的过程中,scRNA-seq的关键技术包括细胞捕获技术和数据处理方法。细胞捕获技术决定了细胞如何被筛选、获取怎样的测序外的补充信息、数据产量。三种最常用的细胞捕获技术是基于微孔、微液流和微滴。数据处理方法决定了如何对scRNA-seq数据进行质控、标准化、降维、聚类、差异分析等。目前比较常用的数据处理方法主要包括Seurat、Monocle、Scanpy、Linnorm等。
技术应用
从动物到植物,从身体机理到疾病机制,scRNA-seq以前所未有的方式准确、快速地分析mRNA信息,可以帮助我们解决各种科学难题。
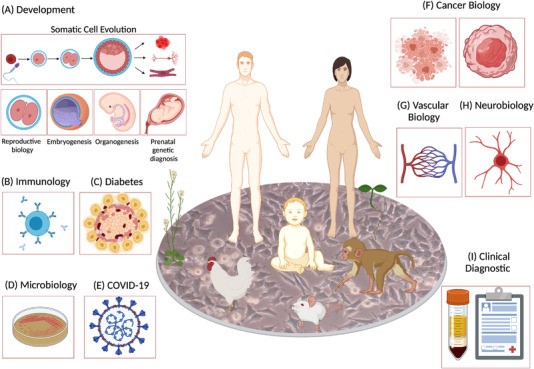
图3. 单细胞测序技术广泛应用于多种领域
传统的批量RNA测序或者微阵列技术分析可能导致稀有细胞群和亚群的丢失,但scRNA-seq的应用能够让我们以单细胞分辨率识别稀有的细胞类型,成为剖析、识别、分类和发现来自不同人体器官和组织的新的或罕见的细胞类型和亚型的有力工具,在发育、免疫学、糖尿病、微生物学、Covid-19、癌症生物学、血管生物学、神经生物学、临床诊断和许多其他学科中提供更深刻的健康和疾病信息。这个优势也使得研究者可以进一步描述组织或者细胞群中的细胞多样性。通过分析不同细胞类型或亚群的组成、分布、功能及相互通讯,创建细胞图谱,提供组织或生物体的细胞组成的全貌。
以癌症研究为例,scRNA-seq 分析可以在肿瘤的不同发育阶段区分功能健康的细胞和癌细胞,也可以分析肿瘤异质性。可以帮助研究肿瘤中存在的不同克隆群体、微环境成分和免疫浸润情况,有助于分析肿瘤形成的过程。此外,也可以通过识别和确定对不同药物的敏感性来进行更精确的预后和诊断,并制定最有效的癌症治疗策略。例如,Pan et al.结合IHC和单细胞测序,在胰腺导管腺癌异种移植模型中检查了CD47阻断的疗效,对CD45+免疫细胞进行了RNA测序(图5)。他们的研究发现CD47诱导胰腺导管腺癌中肿瘤微环境的肿瘤浸润免疫细胞的分区重塑,有助于优化免疫检查点抑制剂疗法。

图4. 单细胞RNA测序研究喉鳞状细胞癌(LSCC)中的肿瘤细胞异质性
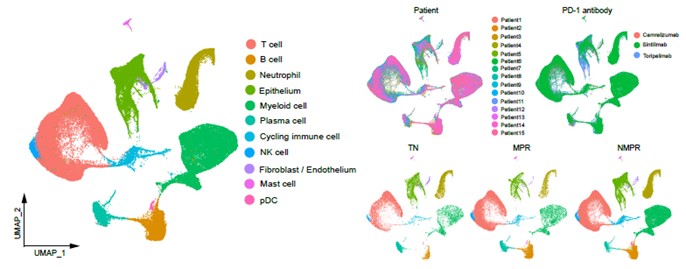 图5. 单细胞RNA测序揭示免疫疗法后的非小细胞肺癌肿瘤微环境重塑
图5. 单细胞RNA测序揭示免疫疗法后的非小细胞肺癌肿瘤微环境重塑
除了研究疾病相关的分子机制,优化药物治疗,scRNA-seq可以帮助研究物种的发育。通常在器官发育早期,细胞集合从组织学上看起来相同,但之后会往不同的方向分化,成为不同的细胞类型。了解这些分化细胞的精准基因表达有助于提高对器官发育过程的理解,单细胞测序便可以确定细胞的分化相关的不同基因表达模式。此外,这个技术可以用来重建不同发育阶段或不同物种之间的单细胞谱系树,定义所有的转录因子、生长因子和受体在每个细胞类型中的表达、揭示发育过程中的分化轨迹、关键转折点和命运决定因素。
在单细胞测序飞速发展的现在,单细胞转录组测序已经被运用于不同的研究领域,成为了研究者的好帮手。我们LabEx携手BD为大家带来多种单细胞测序服务,包括结合单细胞转录组学和单细胞蛋白测序的多组学分析,单细胞免疫组库分析等等。

在下周的文章中,我们将会为大家带来单细胞蛋白测序的介绍,原理及技术应用。
Reference:
Wu, X., Yang, B., Udo-Inyang, I., Ji, S., Ozog, D., Zhou, L., & Mi, Q. S. (2018). Research Techniques Made Simple: Single-Cell RNA Sequencing and its Applications in Dermatology. The Journal of investigative dermatology, 138(5), 1004–1009. https://doi.org/10.1016/j.jid.2018.01.026
Jovic, D., Liang, X., Zeng, H., Lin, L., Xu, F., & Luo, Y. (2022). Single-cell RNA sequencing technologies and applications: A brief overview. Clinical and translational medicine, 12(3), e694. https://doi.org/10.1002/ctm2.694
Tang, F., Barbacioru, C., Wang, Y., Nordman, E., Lee, C., Xu, N., Wang, X., Bodeau, J., Tuch, B. B., Siddiqui, A., Lao, K., & Surani, M. A. (2009). mRNA-Seq whole-transcriptome analysis of a single cell. Nature methods, 6(5), 377–382. https://doi.org/10.1038/nmeth.1315
Islam, S., Kjällquist, U., Moliner, A., Zajac, P., Fan, J. B., Lönnerberg, P., & Linnarsson, S. (2011). Characterization of the single-cell transcriptional landscape by highly multiplex RNA-seq. Genome research, 21(7), 1160–1167. https://doi.org/10.1101/gr.110882.110
Gierahn, T. M., Wadsworth, M. H., 2nd, Hughes, T. K., Bryson, B. D., Butler, A., Satija, R., Fortune, S., Love, J. C., & Shalek, A. K. (2017). Seq-Well: portable, low-cost RNA sequencing of single cells at high throughput. Nature methods, 14(4), 395–398. https://doi.org/10.1038/nmeth.4179
Song, L., Zhang, S., Yu, S., Ma, F., Wang, B., Zhang, C., Sun, J., Mao, X., & Wei, L. (2020). Cellular heterogeneity landscape in laryngeal squamous cell carcinoma. International journal of cancer, 147(10), 2879–2890. https://doi.org/10.1002/ijc.33192
Pan, Y., Lu, F., Fei, Q., Yu, X., Xiong, P., Yu, X., Dang, Y., Hou, Z., Lin, W., Lin, X., Zhang, Z., Pan, M., & Huang, H. (2019). Single-cell RNA sequencing reveals compartmental remodeling of tumor-infiltrating immune cells induced by anti-CD47 targeting in pancreatic cancer. Journal of hematology & oncology, 12(1), 124. https://doi.org/10.1186/s13045-019-0822-6












 沪公网安备31011502400759号
沪公网安备31011502400759号
 营业执照(三证合一)
营业执照(三证合一)


